在先前教學文章中介紹了各式各樣生資工具,
儘管 Alpha 與 Beta 多樣性分析可以一覽組內與組間的特徵,
若想要一窺究竟是哪些菌種在組別間數一數二發光發熱,
LEfSe 就是一款以統計分析為基礎,
在組別間進行比較,
獲得各組別微生物生物標記 (Biomarker)的軟體。
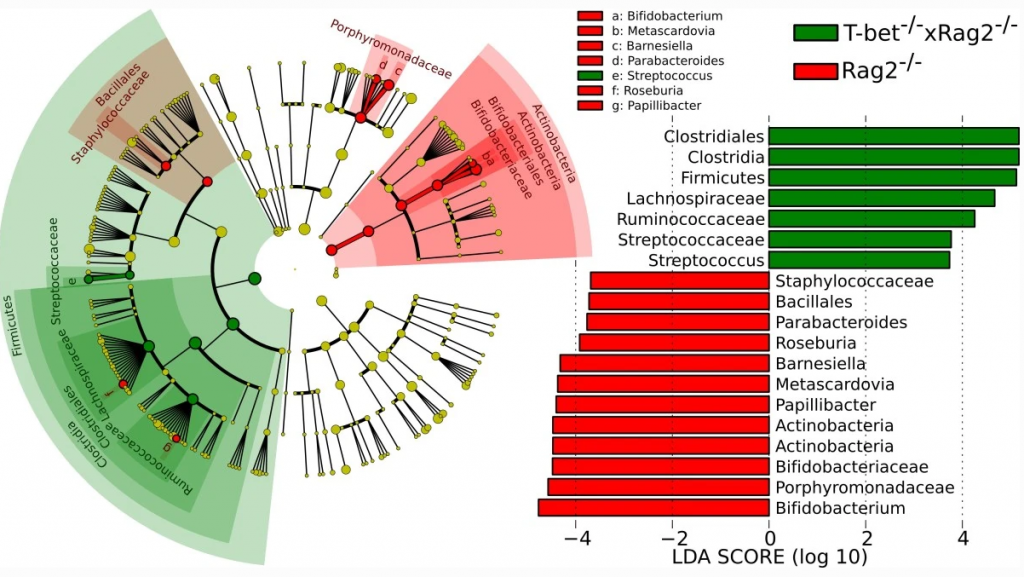
左側由內到外的每一層同心圓分別代表界~種的生物階層,每一點代表一個某階層的名稱,黃色表示無顯著差異,紅綠等顏色則代表該菌種在對應組別具有顯著差異。
右側則為 LDA Score 結果,在本圖中橫軸負數代表偏向紅色組,正數則代表偏向綠色組,量化後絕對值後數值越大代表越顯著。
(Segata, Nicola, et al., 2011)
為方便後續講解我們做個小約定,
在本篇提及的「菌種」二字指的是不同階層的分類名稱,
暫時拋下生物嚴謹定義的種一詞為「種 (species)」層級,
因為我們會提及不同階層,
例如菌物界為一菌種、擬桿菌門為一菌種,
在我找到一個合適的名詞取代之前大家忍耐一下> <
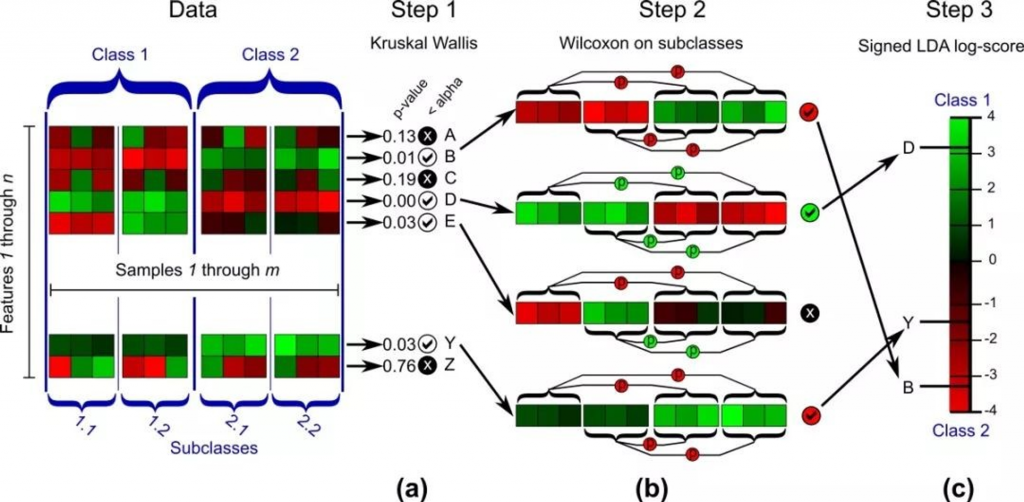
上圖中的a, b, c即對應下列點 1, 2, 3,Class1, Class2即不同組別。
(Segata, Nicola, et al., 2011)
LEfSe 所吃的輸入檔案比 [Day 19] PICRUSt2 所需的檔案格式複雜且傲嬌,
有位偉大的前輩受不了製作輸入檔案的痛苦,
開發了dokdo 套件拯救芸芸眾生。
在啟動 QIIME2 環境的前提下,
安裝 dokdo 套件,幫助製作 LEfSe 所需的輸入檔案。
經測試發現,QIIME2版本需要在2022.2(含)以下才能順利安裝dokdo,
因此本篇文章以QIIME2 2022.2版本教學。
安裝 QIIME2 2022.2
wget https://data.qiime2.org/distro/core/qiime2-2022.2-py38-linux-conda.yml
conda env create -n qiime2-2022.2 --file qiime2-2022.2-py38-linux-conda.yml
啟動 QIIME2 環境
conda activate qiime2-2022.2
安裝 dokdo 套件
conda install -c hcc dokdo
我也不知道為什麼會有 Day 31 ,興致一來就有了。
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
